异染色质是指基因组中用DAPI 染色区域较深,相对不开放的区域。这类区域被认为是基因组中的黑洞。之前研究认为异染色质的基因组通常处于沉默状态。 随着转录组学测序技术的发展,大家发现异染色质并非一直保持沉默。那么这些活跃的异染色质有什么功能呢? 基因组学研究发现细胞间期异染色质的稳定可以保证基因组结构的稳定。分裂期的异染色质,尤其是位于着丝粒区域,以及近着丝粒区域的异染色质, 其稳定性可以帮助遗传信息在细胞分裂中有效的高保真传递。 在细胞分裂过程中,异染色质中的表观marker(H3K9me3)会随机分配到两个子细胞中。随机表观修饰的丢失,会使得异染色质变得不稳定。 那么生命体是如果在体细胞中维持异染色质的稳定呢?
另一方面,重复序列是一类在基因组中存在的多个拷贝的序列,其在基因组,尤其是异染色质中占据着相当大的比例。由于重复序列的多拷贝性质,测序分析中很难准确找到其定位。也由于这个特点,重复序列的相关研究一直被滞后。但是重复序列的功能在一些已知的通路中已经被报道。 而且这些功能多伴随着重复序列的转录。所以系统研究重复序列的转录情况以及这些转录产生的RNA的结合位点将为我们系统解析重复序列的功能打开一扇大门。
基于这一系列问题, 近日,加州大学圣地亚哥分校付向东教授和中国科学院生物物理研究所陈润生院士、何顺民研究员合作,在Genome Research上发表了长文文章"Active retrotransposons help maintain pericentromeric heterochromatin required for faithful cell division"的封面文章[1]。该工作发现了在果蝇S2细胞中,存在一类来自于特定重复序列的RNA,这些RNA显著结合在着丝粒近端组成型异染色质。这种结合可以帮助维持异染色质的状态,进而可以维系细胞进行正确的有丝分裂。
在本研究中,郝亚静等研究人员首先利用付向东组开发的全基因组RNA-染色质相互作用测序技术GRID-seq[2-3], 通过改进计算方法, 第一次系统的构建了果蝇S2细胞中重复序列来源RNA的全基因组结合图谱。进一步的,郝亚静等第一次发现,不同于其他RNA倾向于结合在开放活跃的染色质区域,一类重复序列来源的RNA显著的富集在组成型异染色质区域。 这类RNA随后被命名为CHARRs(组成型异染色质相关RNA)。 CHARRs不仅可以通过cis作用结合到邻近位置,还可以发生trans远距离互作。 有趣的是,在异染色质被激活的区域,如果Local RNA结合多的区域,trans 结合的RNA就少,如果local 结合少的区域,trans结合的RNA就多(图1)。
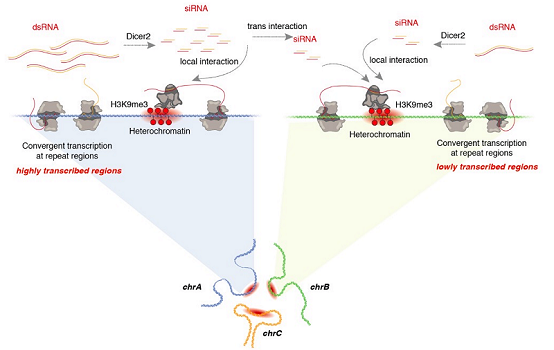
图1:组成型异染色质维持的模式图
该研究还发现CHARRs主要来自活跃的gypsy元件,符合endo-siRNAs特性:长度在~21nt左右,可以被Dcr-2加工成小RNA,并被AGO2结合。 之前的报道包括本文发现敲降Dicer-2会引起异染色质丢失,细胞分裂期染色体排列和分离异常。 但是,人们对这一关键表型到底是由siRNA通路还是Dicer-2在核内的其他功能导致的却知之甚少。郝亚静等首次报道了人工合成的CHARRs来源的endo-siRNA mimics可以恢复由于缺失Dcr-2造成的组成型异染色质修饰(H3K9me3)的缺失以及细胞分裂的异常表型。而且这种恢复作用具有序列特异性。 这是第一次在高等物种中直接证明是由endo-siRNA来调控组成型异染色质的稳定。 也是第一次认识到活跃转座子在基因组结构以及遗传信息稳定传递中的有利效应。 同时这项研究工作也展示了付向东教授课题组开发的GRID-seq技术在系统研究RNA-染色质互作组/图谱及其功能上的强大能力。
加州大学圣地亚哥分校付向东教授和中国科学院生物物理研究所陈润生院士、何顺民研究员为论文的共同通讯作者;生物物理所郝亚静博士(现为加州大学圣地亚哥分校付向东研究组博士后)、王东鹏博士(现为美国国立卫生研究院国家癌症研究所博士后)为论文的共同第一作者。健康大数据研究中心张鹏副研究员、吴树恒博士参与了此项研究。该项目受到国家自然科学基金委、国家重点研发计划、美国国立卫生研究院基金等项目资助。
文章链接:https://genome.cshlp.org/content/30/11/1570.full
参考文献:
[1] Hao, Y., Wang, D., Wu, S., Li, X., Shao, C., Zhang, P., Chen, J.Y., Lim, D.H., Fu, X.D., Chen, R., and He, S. (2020). Active retrotransposons help maintain pericentromeric heterochromatin required for faithful cell division. Genome Res.
DOI: https://doi.org/10.1101/gr.256131.119
[2] Li, X., Zhou, B., Chen, L., Gou, L.T., Li, H., and Fu, X.D. (2017). GRID-seq reveals the global RNA-chromatin interactome. Nat Biotechnol 35, 940-950.
DOI: https://doi.org/10.1038/nbt.3968
[3] Zhou, B., Li, X., Luo, D., Lim, D.H., Zhou, Y., and Fu, X.D. (2019). GRID-seq for comprehensive analysis of global RNA-chromatin interactions. Nat Protoc 14, 2036-2068.
DOI: https://doi.org/10.1038/s41596-019-0172-4
本内容来源于网络,如有侵权请联系本网站删除,谢谢!

