
 敬请关注公众号
敬请关注公众号
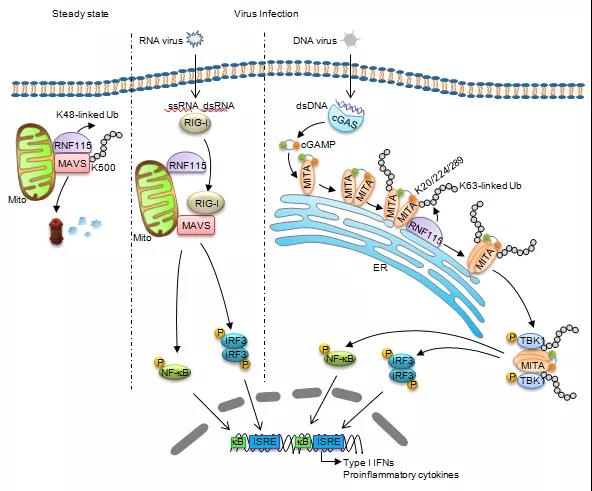
病毒感染与复制的过程中产生的病毒核酸,通过结合宿主细胞内的相关模式识别受体(pattern-recognition receptor)激活一系列的信号级联反应,进而诱导I型干扰素、TNF等细胞因子和促炎因子的表达[1]。MAVS(又叫做VISA)以及MITA(也叫STING)作为重要的接头蛋白,分别介导胞浆中识别RNA的模式识别受体和识别DNA的模式识别受体所介导的信号转导。研究表明,MAVS和MITA的活性与稳定性受到泛素化与去泛素化修饰的严格调控[2]。目前有多个E3泛素连接酶参与病毒感染后MAVS泛素化,进而激活或抑制RNA病毒感染诱导的信号转导,但对于处于静息状态细胞中MAVS的调控及其生理意义尚未见报道。此外,DNA病毒感染诱导MITA发生K63链接的泛素化修饰,促进其寡聚化进而激活下游信号级联反应,但介导这一过程的E3泛素连接酶仍然不清楚[3-8]。
近日,武汉大学医学研究院/生命科学学院钟波教授带领的研究小组在Nature Communications发表了最新研究成果:RNF115 plays dual roles in innate antiviral responses by catalyzing distinct ubiquitination of MAVS and MITA。研究发现了天然免疫过程中,泛素连接酶RNF115调控MAVS和MITA不同时空泛素化以应对病毒感染的机制。

他们发现在静息状态下,E3泛素连接酶RNF115与MAVS持续相互作用,诱导MAVS发生K48链接的泛素化并促进其降解,防止细胞中MAVS的过度累积。在RNA病毒感染后,识别RNA的受体RIG-I通过招募MAVS进而阻断RNF115与MAVS相互作用,激活下游信号级联反应。敲除RNF115的组织和细胞中,MAVS的蛋白水平显著升高。与野生型小鼠相比,RNA病毒感染后RNF115敲除小鼠血清中I型干扰素等细胞因子表达水平升高,小鼠更加抵抗EMCV感染。这些结果表明,RNF115通过泛素修饰MAVS调控静息状态下MAVS的蛋白水平。
随后,该研究组发现DNA病毒感染诱导RNF115在内质网上聚集,并与MITA相互作用。RNF115诱导MITA发生K63链接的泛素化修饰,从而促进MITA的寡聚化、从内质网向ERGIC的迁移以及对下游蛋白激酶TBK1的招募。敲除RNF115显著抑制HSV-1感染诱导的I型干扰素的表达,RNF115敲除小鼠对HSV-1更加易感。有趣的是,研究者发现敲除RNF115并不影响病毒感染诱导MITA的二聚化,暗示MITA通过“二聚化-泛素化-寡聚化-迁移” 四个顺序发生的步骤而活化。
该研究阐明了E3泛素连接酶RNF115调控RNA和DNA病毒感染诱导I型干扰素表达的分子机制,同时揭示了MAVS与MITA在病毒感染的不同时空发生泛素化修饰的新机制,推动了我们对抗病毒天然免疫反应机理的理解,为未来开发抗病毒药物提供理了论基础。
据悉,武汉大学生命科学学院2018级博士研究生张志东和医学研究院2018级博士研究生熊天晨为该论文的共同第一作者。钟波教授是本研究的通讯作者。
原文链接:https://doi.org/10.1038/s41467-020-19318-3
参考文献
[1] Hu MM & Shu HB (2018) Cytoplasmic Mechanisms of Recognition and Defense of Microbial Nucleic Acids. Annu Rev Cell Dev Bi 34:357-379.
[2] Li K & Zhong B (2018) Regulation of Cellular Antiviral Signaling by Modifications of Ubiquitin and Ubiquitin-like Molecules. Immune Netw 18(1).
[3] Ni GX, Konno H, & Barber GN (2017) Ubiquitination of STING at lysine 224 controls IRF3 activation. Sci Immunol 2(11).
[4] Seo GJ, et al. (2018) TRIM56-mediated monoubiquitination of cGAS for cytosolic DNA sensing. Nat Commun 9.
[5] Tsuchida T, et al. (2010) The Ubiquitin Ligase TRIM56 Regulates Innate Immune Responses to Intracellular Double-Stranded DNA. Immunity 33(5):765-776.
[6] Yang Q, et al. (2017) TRIM32-TAX1BP1-dependent selective autophagic degradation of TRIF negatively regulates TLR3/4-mediated innate immune responses. Plos Pathog 13(9).
[7] Ye LY, et al. (2019) USP49 negatively regulates cellular antiviral responses via deconjugating K63-linked ubiquitination of MITA. Plos Pathog 15(4).
[8] Zhang J, Hu MM, Wang YY, & Shu HB (2012) TRIM32 Protein Modulates Type I Interferon Induction and Cellular Antiviral Response by Targeting MITA/STING Protein for K63-linked Ubiquitination. J Biol Chem 287(34):28646-28655.
本内容来源于网络,如有侵权请联系本网站删除,谢谢!