
 敬请关注公众号
敬请关注公众号

植物生长素是一类可以调节植物生长的植物激素,生长素响应因子(Auxin response factor, ARF)则是一类特异结合于生长素响应基因启动子上从而发挥激活或抑制作用的转录因子,其对生长素合成的调控影响到植物生长发育的各个阶段。
1. 基本信息
Locus:AT1G30330
Name::ARF6
2. 基因背景介绍
生长素响应因子(Auxin Response Factor)是生长素信号通路中参与植物不同组织生长和发育的转录因子。目前,ARF家族在拟南芥中一共鉴定到37名成员。之前的研究表明,ARF家族在植物的根发育、花成熟、叶片成长以及响应逆境等方面发挥了重要的作用。ARF6作为ARF家族的重要成员,也参与到了雄蕊生长以及花成熟等过程中。并且和ARF8存在功能冗余的现象,该现象是ARF家族中唯一存在的情况[1]。
3. 突变体信息
ARF6的突变体的表型为花器官的数量和位置正常,但是可能无法形成正常的果荚。部分原因可能是花丝伸长和花药开裂稍微延迟,导致花药距离柱头太远,因而植株自花授粉效率就变低。在国内拟南芥突变体共享中心AraShare中有一个该基因的T-DNA插入突变体(N684361,详情请查阅 http://www.arashare.cn/)。

图1 arf 6突变体生长情况
4. 基因表达谱
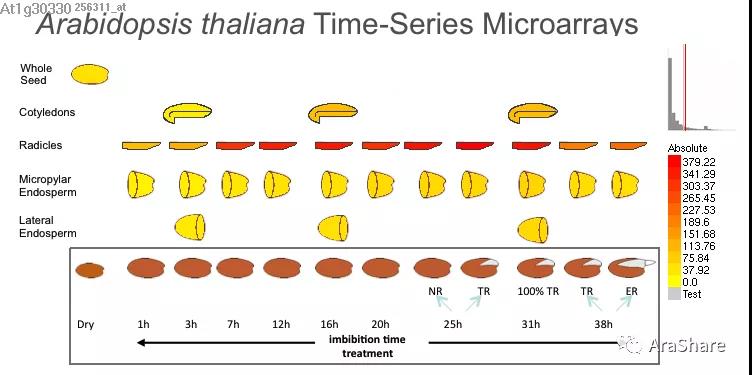
图2. 拟南芥中ARF6基因表达谱
如图所示,ARF6在拟南芥干旱处理7h及以上的根部表达量较高,在31h后开始下降。在整个种子、子叶以及胚乳中的表达量都不及根部的高。
5. 研究历史与进展简述
1999年,在前人利用克隆技术鉴定ARF1基因之后,Guilfoyle团队趁热打铁利用当时的“宠儿”-DNA探针技术鉴定了ARF其余家族成员,这其中就包括了ARF6基因。紧接着,他们又对ARF6的结构域进行了探究。研究者用ARF6的启动子质粒与P3(4X)启动子或大豆GH3启动子融合的生长素应答的GUS报告质粒进行了测试。在没有生长素的情况下,P3(4X)报告基因的基础活性较低,并被生长素诱导了约20倍。与P3(4X)报告基因相比,GH3报告基因具有更高的基础活性,但仍被生长素诱导5倍以上。该结果表明ARF6具有激活转录的作用。并且为了验证这一结果,研究者构建了删除了DBD和MR的ARF蛋白的氨基端,结果ARF6启动子对转录的激活程度为3-6倍,大大低于含有Q-rich MR的ARF6所产生的激活效应[2]。该结果也再一次表明含有Q-rich MR的ARF转录因子一般具有激活转录的作用。而具有转录激活作用的ARF6在生长发育中具体行使了什么功能呢?为一探究竟,Guilfoyle团队于2005年进行了突变实验。结果表明当分别敲除掉arf6和arf8单突变体和半突变(其中一个突变为纯合子,另一个突变为杂合子)延缓了雄蕊发育,降低了繁殖力,说明ARF6和ARF8基因的表达量影响了花成熟的时间。相对应的,双突变(arf6和arf8)的实验结果表明改变了生长素响应基因的表达,导致花具有不育的闭合花芽,短花瓣和短雄蕊花丝以及雌蕊成熟的延缓。然而,整个花的生长素水平在花的成熟过程中并没有发生变化,这说明生长素可能只在特定的环境条件下或在花的局部器官或组织中调控花的成熟[1]。
2012年,牛津大学的Ce´dric Finet等人在MOLECULARBIOLOGY AND EVOLUTION在线发表了一篇ARF家族进化的文章,在前人对ARF家族系统进化研究的基础上,对ARF家族进行了可变剪切和基因组截断的注释。结果显示ARF6和ARF8都仅发生了可变剪切,而未有基因组截断的情况发生。这一结果也与之前的功能验证结果相辅相成,再次证明了ARF6和ARF8是一对难舍难分的姐妹对[3]。
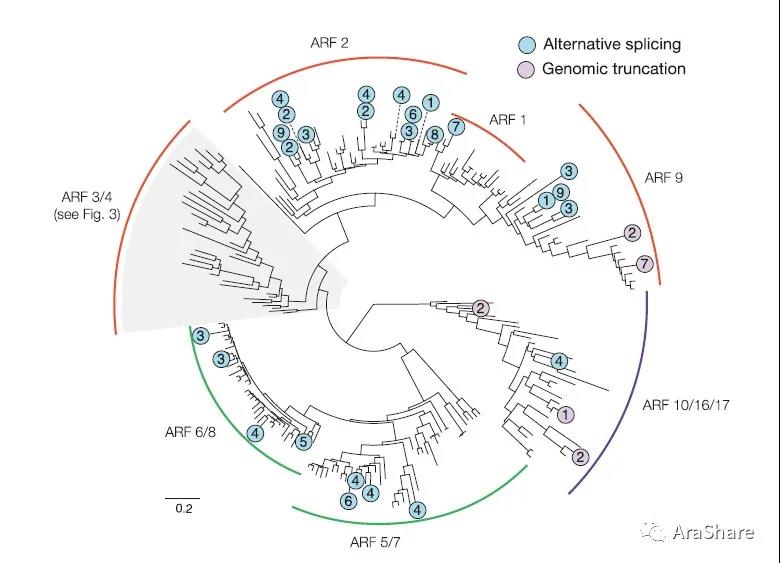
图3. 拟南芥中ARF家族系统进化树
6. 基因网络
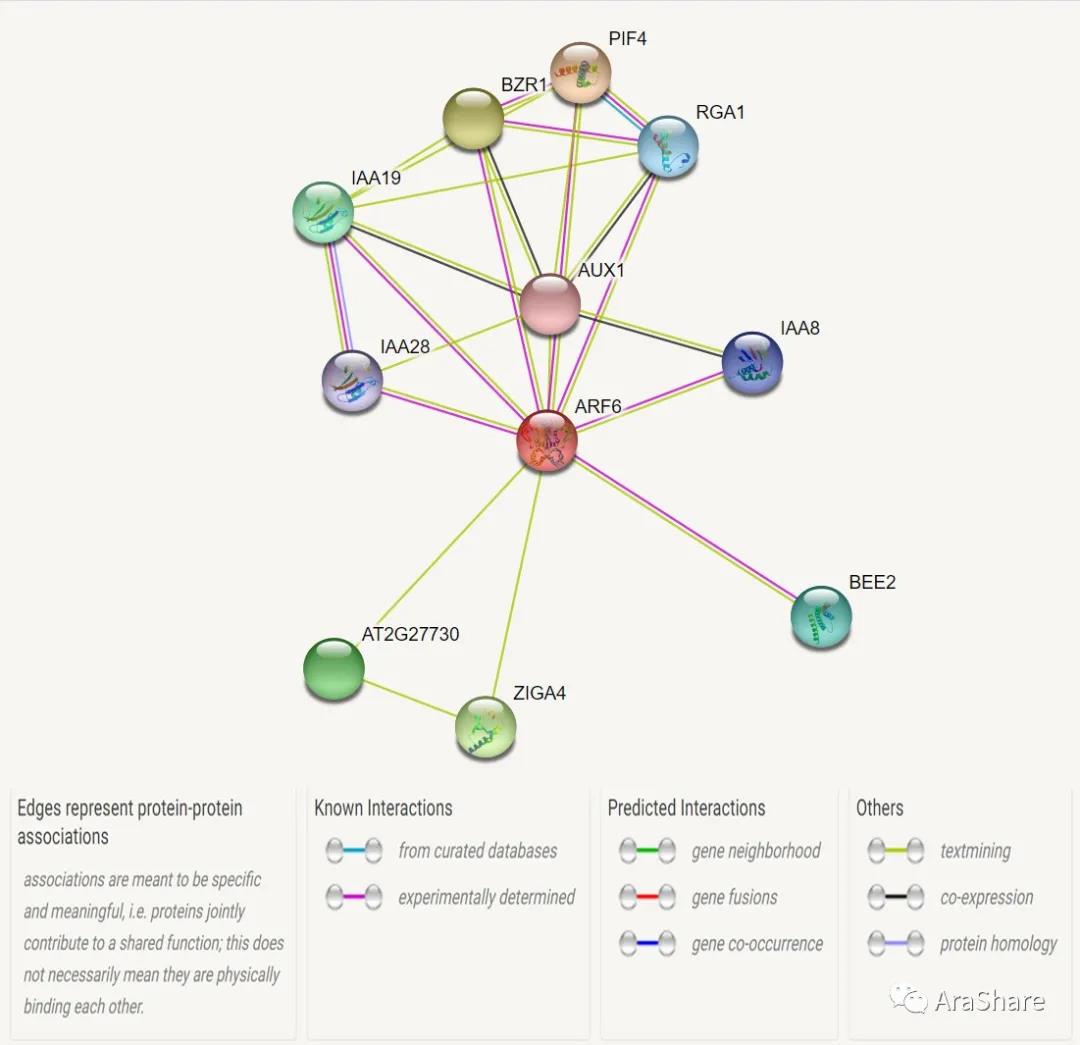
图4.STRING 预测ARF6蛋白互作网络
如图所示,ARF6和图中所有基因都是基因邻接关系,和PIF4、IAA19、IAA28、BZR1、RGA1、AUX1、BEE2以及IAA8呈现的是基因融合的关系,其中IAA19、AUX1、BZR1以及IAA1存在共表达的关系。
7. 其他物种研究情况简介
随着测序技术的发展,越来越多物种基因组的完成,ARF家族在杨树[4]、水稻[5]、小麦[6]以及玉米[7]中的鉴定已经完成。包括在黄瓜[8]、木瓜[9]以及桃[10]中也完成了ARF家族的鉴定。其中在杨树中的实验结果表明PeARF6可以参与不定根的发育。这一结果和大部分研究者将焦点聚集在花的成熟上不一样,而是从不定根的角度去探究了ARF6在生长发育中发挥的作用,为ARF家族的研究提供了新的视角。
8. 展望
虽然越来越多物种的ARF家族已经被鉴定,但是不同物种因为进化过程的不保守性可能存在着差异,尤其是被子植物和裸子植物生殖发育存在较大差异,而ARF6基因和生殖发育又息息相关,因而还需要进一步的实验去验证ARF6在不同物种中的功能。
参考文献
[1] P. Nagpal, C.M. Ellis, H. Weber, S.E. Ploense, L.S.Barkawi, T.J. Guilfoyle, G. Hagen, J.M. Alonso, J.D. Cohen, E.E. Farmer, J.R.Ecker, J.W. Reed, Auxin response factors ARF6 and ARF8 promote jasmonic acidproduction and flower maturation, DEVELOPMENT, 132 (2005) 4107-18.
[2] Activation andRepression of Transcription by Auxin-Response Factors Author(s): Tim Ulmasov,Gretchen Hagen and Tom J. Guilfoyle Source: Proceedings of the National Academyof Sciences of the United States of America, Vol. 96, No. 10 (May 11, 1999),pp. 5844-5849[J].
[3] C. Finet, A. Berne-Dedieu, C.P. Scutt, F. Marlétaz, Evolution of the ARF Gene Family in Land Plants: Old Domains, New Tricks, MOL BIOL EVOL, 30 (2013) 45-56.
[4] C. Yang, M. Xu, L. Xuan, X. Jiang, M. Huang,Identification and expression analysis of twenty ARF genes in Populus, GENE,544 (2014) 134-144.
[5] D. Wang, K.Pei, Y. Fu, Z. Sun, S. Li, H. Liu, K. Tang, B. Han, Y. Tao, Genome-wideanalysis of the auxin response factors (ARF) gene family in rice (Oryzasativa), GENE, 394 (2007) 13-24.
[6] L. Qiao, W. Zhang, X. Li, L. Zhang, X. Zhang, X. Li,H. Guo, Y. Ren, J. Zheng, Z. Chang, Characterization and Expression Patterns ofAuxin Response Factors in Wheat, FRONT PLANT SCI, 9 (2018).
[7] H. Xing, R.N. Pudake, G. Guo, G. Xing, Z. Hu, Y.Zhang, Q. Sun, Z. Ni, Genome-wide identification and expression profiling ofauxin response factor (ARF) gene family in maize, BMC GENOMICS, 12 (2011)178-178.
[8] S.Q. Liu, L.F. Hu, Genome-wide analysis of the auxinresponse factor gene family in cucumber, Genet Mol Res, 12 (2013) 4317-31.
[9] K. Liu, C. Yuan, H. Li, W. Lin, Y. Yang, C. Shen, X.Zheng, Genome-wide identification and characterization of auxin response factor(ARF) family genes related to flower and fruit development in papaya (Caricapapaya L.), BMC GENOMICS, 16 (2015).
[10] D. Diao, X. Hu, D. Guan, W. Wang, H. Yang, Y. Liu,Genome-wide identification of the ARF (auxin response factor) gene family inpeach and their expression analysis, MOL BIOL REP, 47 (2020) 4331-4344.
本推文基于科学文献和网络资讯写作而成,未经领域内专家审阅;限于笔者学识水平,难免存在错漏,仅供参考!